3 月 7 日,同济大学附属同济医院王晨飞课题组在 Nucleic Acids Research 杂志上发表文章 STRIDE: accurately decomposing and integrating spatial transcriptomics using single-cell RNA sequencing,开发了空间转录组与单细胞整合分析工具 STRIDE,通过机器学习方法及数据整合,将空间转录组学数据提升至单细胞精度。

高通量单细胞测序技术的发展,使得研究人员能够在单细胞分辨率研究复杂生物系统中细胞的异质性,但在实验过程中需要经历组织消化等操作,导致细胞在组织中原始位置信息的丢失。新兴的空间转录组学技术则在定量基因表达水平的同时,保留了细胞的空间信息,研究人员得以在空间背景下研究细胞不同的功能状态以及细胞间的相互作用。然而,由于技术的限制,目前大多数空间转录组或是检测的基因数目较少,或是无法达到单细胞的分辨率,这些都为空间转录组的高精度分析带来了巨大的挑战。
STRIDE(Spatial transcriptomics deconvolution by topic modeling)基于主题模型这一机器学习算法,旨在借助匹配的单细胞数据对空间转录组进行细胞类型的解析。STRIDE 首先从单细胞数据中学习训练出细胞类型相关的主题模型,接着将该主题模型应用于空间转录组数据中,以获得每一个空间位置上的主题分布,从而推测其精确的细胞组成。此外,STRIDE 还提拱了包括空间特征识别及可视化、空间聚类以及三维组织重建在内的下游分析功能。
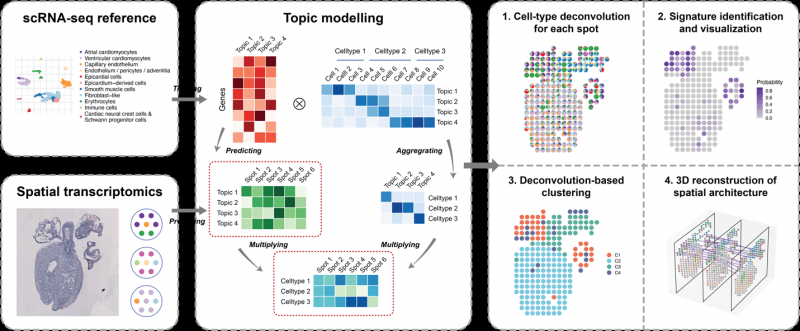
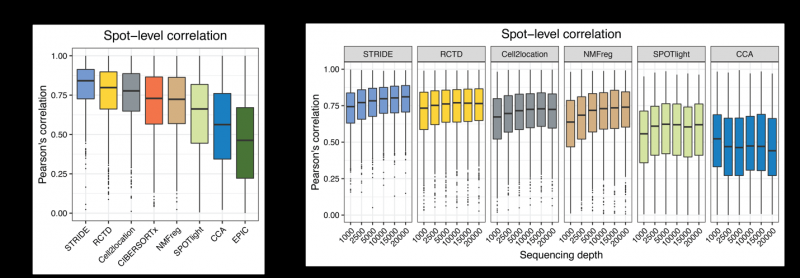
为了论证 STRIDE 在解析细胞组成比例上的准确性,文章从乳腺癌单细胞数据中模拟产生了空间转录组数据,并对其进行细胞组成的预测,与其他已经发表的空间转录组反卷积分析工具相比,STRIDE 有着更高的准确性与灵敏度,且在解析空间上有共定位关系的细胞组成以及测序深度较低时具有更好的表现。
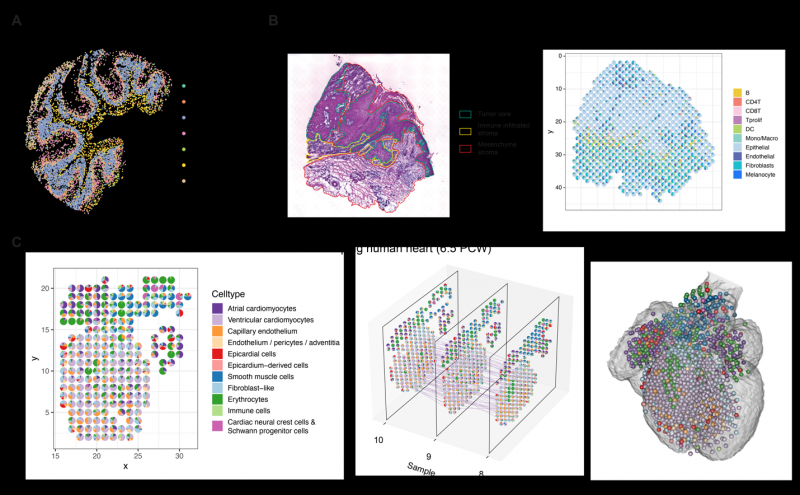
为了进一步论证 STRIDE 可被广泛用于不同组织、不同技术的空间转录组数据的分析中,文章将其分别应用于三套来源于不同生物学系统的数据,包括小鼠小脑的 Slide-seq 数据、人类鳞状细胞癌 Spatial Transcriptomics 数据、人胚胎心脏发育 10X Visium 数据。在小鼠小脑数据的分析中,STRIDE 不仅准确地构建了小鼠小脑的空间细胞图谱,还发现了主题特征比已知标记基因能够更好定义细胞的空间分布。
在人类鳞状细胞癌的应用中,STRIDE 解析出肿瘤微环境中各种细胞类型的空间分布,在此基础上结合空间邻近信息进行了空间聚类分析,并利用单细胞数据对空间转录组做了精度提升,发现肿瘤内部和肿瘤边界的恶性细胞具有不同的特征及功能。在人胚胎心脏发育数据中,STRIDE 对三个不同发育时期的心脏切片进行了细胞类型的注释,准确定位了一种稀有的细胞类型,接着基于连续切片之间「主题」分布的相似性,以及空间结构的相似性,STRIDE 进行了连续切片数据间的整合分析,并成功构建出发育阶段心脏的三维模型。
总体来说,STRIDE 是一种基于主题模型的空间转录组分析工具,可以灵活应用于各种生物系统,同时也支持各种主流的空间转录组技术,能够将其提升至单细胞精度。同济大学附属同济医院王晨飞研究员、附属一妇婴保健院吴秋博士为该论文共同通讯作者,博士研究生孙冬青为第一作者。该项工作得到了国家自然科学基金委及上海市科委等项目的重要支持。
课题组聚焦单细胞及空间多组学数据机器学习方法开发,并将其应用于解决肿瘤免疫微环境、胚胎发育过程中表观修饰及空间信息的异质性,探索其对细胞状态改变及命运决定的调控机制。
好文章,需要你的鼓励